Informatics Educational Institutions & Programs
| Alphasatellitidae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

Virion der Geminialphasatellitinae (links), | ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Alphasatellitidae | ||||||||||||||
| Links | ||||||||||||||
Die Alphasatellitidae sind eine Familie Satellitenviren, die zu ihrer Replikation auf ein Helfervirus angewiesen sind. Ihr Genom ist ein einzelnes zirkuläres Einzelstrang-DNA-Molekül. Sie werden auch informell als Alphasatelliten oder kurz α-Satelliten bezeichnet.[2] Die ersten Alphasatelliten wurden 1999 beschrieben und mit den Viren der Baumwollblattroll- und der Gelbaderkrankheit bei Ageratum-Pflanzen (Cotton leaf curl / Ageratum yellow vein disease) in Verbindung gebracht.[Anm. 1] Diese Viren gehören alle der Gattung Begomovirus aus der Familie Geminiviridae an.[3][4] Seitdem Begomoviren auf molekularer Ebene charakterisiert wurden, wird auch eine zunehmende Zahl von Alphasatelliten beschrieben.
Innerhalb der Familie Alphasatellitidae werden derzeit (Juni 2021) drei Unterfamilien unterschieden: Geminialphasatellitinae (mit Helferviren aus der Familie Geminiviridae), Nanoalphasatellitinae (mit Helferviren aus der Familie Nanoviridae) und Petromoalphasatellitinae (mit Helferviren aus der Familie Metaxyviridae)[1][5]
Die Petromoalphasatellitinae sind 2021 dazu gekommen; ihr Name leitet sich ab von perennial tropical monocotyledons. Die Familie Metaxyviridae ist eine Schwesterfamilie der Nanoviridae und ihre Helferviren rekrutieren sich nach derzeitiger Kenntnis einzig aus der Spezies Coconut foliar decay virus.[6] Alle Helferviren gehören dem Phylum Cressdnaviricota von CRESS-DNA-Viren an.[1]
Diese Viren wurden früher als DNA1-Komponenten (DNA 1 components) bezeichnet.[7][8] Aplhasatelliten wurden zunächst (vor allem) in der Alten Welt gefunden. Inzwischen wurden aber auch mehrere aus der Neuen Welt isoliert, ihre Verbindung mit potentiellen Helferviren muss jedoch oft noch genauer untersucht werden.
Beschreibung
Da das Virus-Genom der Alphasatellitidae im Kapsidprotein der Helferviren verpackt wird, gibt es keine Virionen mit alphasatellitidae-spezifischer Morphologie.
Zudem benötigen Alphasatellitidae, die mit Begomoviren assoziiert sind (Unterfamilie Geminialphasatellitinae), diese Helferviren auch für die Fortbewegung in den Pflanzen (den Wirten der Begomoviren), und die Übertragung durch Insekten (als Vektoren). Sie sind lediglich zur Reproduktion ihres Genoms in den Wirtspflanzen fähig. Sie scheinen selbst keine Krankheiten in Pflanzen zu verursachen; möglicherweise sind sie in der Lage, den Schweregrad einer Infektion durch die Begomoviren zu verringern (in Art der Virophagen). Abgesehen davon scheinen sie den Verlauf der Infektion durch die Begomoviren nicht zu verändern.[9][10]
Alphasatelliten sind auch in Verbindung mit Nanoviridae beschrieben worden (Unterfamilie Nanoalphasatellitinae). Das von den Nanoviridae „geborgte“ Kapsid ist wie bei diesen von ikosaedrischer Geometrie und klein (Größe etwa 12 nm). Aufgrund des segmentierten Genoms der Nanoviridae wurden diese zunächst nicht als eigenständige Genome erkannt.[11][12][13]
Genom

Das Genom der Alphasatellitidae ist zwischen 1300 und 1400 nt (Nukleotiden) lang und weist drei konservierte Merkmale auf:[14]
- eine Haarnadelstruktur (en. hairpin structure),
- einen einzelnen offenen Leserahmen (en. Open reading Frame (ORF)) und
- eine adeninreiche Region.
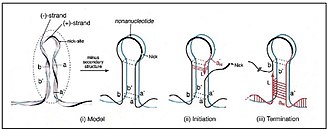
Die Haarnadelstruktur ist eine Gemeinsamkeit mit den Cressdnaviricota, zu denen die Helfeviren gehören. Sie hat eine Schleife, die das Nanonukleotid TAGTATTAC enthält. Dieses Nonanukleotid ist auch allen Nanoviidae gemeinsam und unterscheidet sich von der TAATATTAC-Sequenz der Geminiviridae nur in einem einzigen Nukleotid. Wie bei allen Cressdnaviricota folgt die Genom-Replikation dem Modell der sog. Rolling-Circle-Replikation (RC-Replikation). Die (auch als stem loop, Stammschleife) bezeichnete Haarnadelstruktur enthält den Replikationsursprung (Ori) und wird durch das Replikationsinitiatorprotein (REP oder Rep) eingekerbt, um die virale DNA-Replikation zu starten. Anhand der Haarnadelstrukturen können Alphasatelliten in noch kleinere Kladen (Gattungen) unterteilt werden.[8]
Da der offene Leserahmen (ORF) für ein den Nanoviridae ähnliches Rolling-Circle-Replikationsinitiatorprotein (RC-Rep) kodiert, wird er auch als rep-Gen bezeichnet. Das REP-Protein hat ein Molekulargewicht von 32–37 kDa (Kilo-Dalton) mit ~320 Aminosäuren. Es ist hoch konserviert mit 86,3–100,0 % Aminosäuresequenzidentität zwischen den Isolaten.
Unmittelbar nach dem rep-Gen (dem Gen für das Rep-Protein) befindet sich im Genom eine adeninreiche Region mit einer Länge von ca. 153–169 nt und einem Adenin-Gehalt zwischen 52,3 und 58,4 %. Vermöge phylogenetischer Analyse dieser Region lassen sich Kladen der Alphasatellitidae identifizieren, die denen entsprechen, die bei der phylogenetischen Analyse des gesamten Genoms gefunden wurden.[8] Dieser Teil des Genoms scheint redundant zu sein.[15]
Im Jahr 2010 haben G. Romay und Kollegen von einem mutmaßlichen Mitglied der Alphasatellitidae mit der vorgeschlagenen Bezeichnung Melon chlorotic mosaic virus alphasatellite 1 (MeCMVa1) und dem Helfervirus Melon chlorotic mosaic virus (MeCMV) aus der Gattung Begomovirus berichtet. Im Genom von MeCMVa1 gibt es mutmaßlich einen zweiten ORF. Die Bedeutung dieses Befundes (falls überhaupt vorhanden) war zum Zeitpunkt der Veröffentlichung noch nicht bekannt.[16]
Obwohl das Genom der Alphasatellitidae unsegmentiert ist, scheint es Rekombinationserignisse sowohl unter den Satelliten (als auch ihren Helferviren) zu geben.[17]
Anwendung
Alphasatelliten wurden bei der Entwicklung von Studien zum viralen Gen-Silencing eingesetzt.[18][19]
Evolution

Angesichts der Ähnlichkeiten zwischen den Rep-Proteinen der Alphasatelliten und der Nanoviridae ist es wahrscheinlich, dass sich die Alphasatelliten aus den Nanoviridae entwickelt haben.[8] Dies legt nahe, dass die Alphasatellitidae in der Umgebung der Nanoviridae,[20] vielleicht in der sie enthaltenden Ordnung Mulpavirales anzusiedeln sind. Die Genom-Architektur macht eine Zugehörigkeit zum Phylum Cressdnaviricota wahrscheinlich,[21] ist aber vom ICTV derzeit (24. Juni 2021) noch nicht offiziell bestätigt. Um diese Fragen zu klären, sind weitere Arbeiten in diesem Bereich erforderlich.
Systematik
Satellitenviren können seitens des International Committee on Taxonomy of Viruses (ICTV) mit eigenen Namensendungen versehen werden (unterschiedlich zu denen für normale Viren): -satellitidae für Familien, -satellitinae für Unterfamilien und -satellite für Gattungen (und darunter).
Die Alphasatelliten werden taxionomisch in der Familie Alphasatellitidae zusammengefasst. Diese Familie hat derzeit (Stand 24. Juni 2021) offiziell bestätigt drei Unterfamilien, 18 Gattungen und 85 Arten. Die folgenden Unterfamilien, Gattungen und Spezies sind anerkannt:[1][22]
Familie: Alphasatellitidae
- Unterfamilie: Geminialphasatellitinae – Helferviren: Geminiviridae
- Gattung: Ageyesisatellite (2 Arten)
- Spezies: Ageratum yellow vein Singapore alphasatellite
- Spezies: Cotton leaf curl Saudi Arabia alphasatellite
- Gattung: Clecrusatellite (15 Arten)
- Spezies: Ash gourd yellow vein mosaic alphasatellite
- Spezies: Capsicum India alphasatellite
- Spezies: Chiapas weed alphasatellite
- Spezies: Cleome leaf crumple alphasatellite
- Spezies: Croton yellow vein mosaic alphasatellite
- Spezies: Euphorbia yellow mosaic alphasatellite
- Spezies: Melon chlorotic mosaic alphasatellite
- Spezies: Sida Cuba alphasatellite
- Spezies: Tomato leaf curl Anand alphasatellite
- Spezies: Tomato leaf curl New Delhi alphasatellite
- Spezies: Tomato leaf curl Virudhunagar alphasatellite
- Spezies: Tomato yellow spot alphasatellite
- Spezies: Tomato yellow spot alphasatellite 2
- Spezies: Whitefly associated Guatemala alphasatellite 2
- Spezies: Whitefly associated Puerto Rico alphasatellite 1
- Gattung: Colecusatellite (27 Arten)
- Spezies: Ageratum enation alphasatellite
- Spezies: Ageratum enation alphasatellite
- Spezies: Ageratum yellow vein alphasatellite
- Spezies: Ageratum yellow vein China alphasatellite
- Spezies: Ageratum yellow vein India alphasatellite
- Spezies: Bhendi yellow vein alphasatellite
- Spezies: Cassava mosaic Madagascar alphasatellite
- Spezies: Chilli leaf curl alphasatellite
- Spezies: Cotton leaf curl Egypt alphasatellite
- Spezies: Cotton leaf curl Gezira alphasatellite
- Spezies: Cotton leaf curl Lucknow alphasatellite
- Spezies: Cotton leaf curl Multan alphasatellite
- Spezies: Gossypium darwinii symptomless alphasatellite
- Spezies: Malvastrum yellow mosaic alphasatellite
- Spezies: Malvastrum yellow mosaic Cameroon alphasatellite
- Spezies: Pedilanthus leaf curl alphasatellite
- Spezies: Sida leaf curl alphasatellite
- Spezies: Sida leaf curl alphasatellite 2
- Spezies: Sida yellow vein Vietnam alphasatellite
- Spezies: Sunflower leaf curl Karnataka alphasatellite
- Spezies: Synedrella leaf curl alphasatellite
- Spezies: Tobacco curly shoot alphasatellite
- Spezies: Tomato leaf curl Buea alphasatellite
- Spezies: Tomato leaf curl Cameroon alphasatellite
- Spezies: Tomato leaf curl Pakistan alphasatellite
- Spezies: Tomato yellow leaf curl China alphasatellite
- Spezies: Tomato yellow leaf curl Thailand alphasatellite
- Spezies: Tomato yellow leaf curl Yunnan alphasatellite
- Gattung: Draflysatellite (1 Art)
- Spezies: Dragonfly associated alphasatellite
- Gattung: Gosmusatellite (8 Arten)
- Spezies: Cotton leaf curl Cameroon alphasatellite
- Spezies: Eclipta yellow vein alphasatellite
- Spezies: Gossypium mustelinum symptomless alphasatellite
- Spezies: Hollyhock yellow vein alphasatellite
- Spezies: Mesta yellow vein mosaic alphasatellite
- Spezies: Okra enation leaf curl alphasatellite
- Spezies: Okra yellow crinkle Cameroon alphasatellite
- Spezies: Vernonia yellow vein Fujian alphasatellite
- Gattung: Somasatellite (1 Art)
- Spezies: Sorghum mastrevirus associated alphasatellite
- Gattung: Whiflysatellite (1 Art)
- Spezies: Whitefly associated Guatemala alphasatellite 1
- Unterfamilie: Nanoalphasatellitinae – Helferviren: Nanoviridae
- Gattung: Clostunsatellite (5 Arten)
- Spezies: Milk vetch dwarf alphasatellite 2
- Spezies: Pea necrotic yellow dwarf alphasatellite 2
- Spezies: Sophora yellow stunt alphasatellite 4
- Spezies: Sophora yellow stunt alphasatellite 5
- Spezies: Subterranean clover stunt alphasatellite 2
- Gattung: Fabenesatellite (1 Art)
- Spezies: Faba bean necrotic yellows alphasatellite 2
- Gattung: Milvetsatellite (1 Art)
- Spezies: Milk vetch dwarf alphasatellite 3
- Gattung: Mivedwarsatellite (7 Arten)
- Spezies: Faba bean necrotic stunt alphasatellite
- Spezies: Milk vetch dwarf alphasatellite 1
- Spezies: Milk vetch dwarf China alphasatellite
- Spezies: Parsley severe stunt alphasatellite 3
- Spezies: Parsley severe stunt alphasatellite 4
- Spezies: Pea necrotic yellow dwarf alphasatellite 1
- Spezies: Sophora yellow stunt alphasatellite 2
- Gattung: Sophoyesatellite (2 Arten)
- Spezies: Cow vetch latent alphasatellite
- Spezies: Sophora yellow stunt alphasatellite 3
- Gattung: Subclovsatellite (4 Arten)
- Spezies: Faba bean necrotic yellows alphasatellite 1
- Spezies: Faba bean necrotic yellows alphasatellite 3
- Spezies: Sophora yellow stunt alphasatellite 1
- Spezies: Subterranean clover stunt alphasatellite 1
- Unterfamilie: Petromoalphasatellitinae[6] – Helferviren: Metaxyviridae
- Gattung: Babusatellite (1 Art, früher Nanoalphasatellitinae)
- Spezies: Banana bunchy top alphasatellite 1
- Gattung: Cocosatellite (4 Arten)
- Spezies: Coconut foliar decay alphasatellite 1
- Spezies: Coconut foliar decay alphasatellite 2
- Spezies: Coconut foliar decay alphasatellite 4
- Spezies: Coconut foliar decay alphasatellite 5
- Gattung: Coprasatellite (1 Art)
- Spezies: Coconut foliar decay alphasatellite 7
- Gattung: Kobbarisatellite (1 Art)
- Spezies: Coconut foliar decay alphasatellite 3
- Gattung: Muscarsatellite (3 Arten)
- Spezies: Banana bunchy top alphasatellite 2
- Spezies: Banana bunchy top alphasatellite 3
- Spezies: Cardamom bushy dwarf alphasatellite
Siehe auch
Anmerkungen
- ↑ D. h. mit nicht Arten von Cotton leaf curl virus einerseits und Ageratum yellow vein virus (AYVV) andererseits.
Einzelnachweise
- ↑ a b c d ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, März 2021 (MSL #36)
- ↑ ZKBS: Stellungnahme der ZKBS zur Risikobewertung von nachfolgend aufgelisteten Vertretern der Familie der Geminiviridae als Spender- und Empfängerorganismen für gentechnische Arbeiten gemäß § 5 Absatz 1 GenTSV: …, Az.: 6790-10-98, September 2010
- ↑ Keith Saunders, John Stanley: A nanovirus-like DNA component associated with yellow vein disease of Ageratum conyzoides: evidence for interfamilial recombination between plant DNA viruses. In: Virology. 264. Jahrgang, Nr. 1, November 1999, S. 142–152, doi:10.1006/viro.1999.9948, PMID 10544139.
- ↑ Shahid Mansoora, Sultan H. Khana, Aftab Bashir et al.: Identification of a novel circular single-stranded DNA associated with cotton leaf curl disease in Pakistan. In: Virology. 259. Jahrgang, Nr. 1, Juni 1999, S. 190–199, doi:10.1006/viro.1999.9766, PMID 10364503.
- ↑
- ↑ a b Bruno Gronenborn, Arvind Varsani, J. W. Randles, H. J. Vetten, J. E. Thomas: Create a new subfamily (Petromoalphasatellitinae) with four new genera and six new species (Alphasatellitidae) (ZIP: docx, xlsx), Vorschlag 2020.001P.R.Petromoalphasatellitinae_nsf an das ICTV (angenommen)
- ↑ J. Stanley: Subviral DNAs associated with geminivirus disease complexes. In: Vet. Microbiol. 98. Jahrgang, Nr. 2, Februar 2004, S. 121–9, doi:10.1016/j.vetmic.2003.10.005, PMID 14741124.
- ↑ a b c d Yan Xie, Peijun Wu, Pei Liu, Huanran Gong, Xueping Zhou: Characterization of alphasatellites associated with monopartite begomovirus/betasatellite complexes in Yunnan, China. In: Virol. J. 7. Jahrgang, 2010, S. 178, doi:10.1186/1743-422X-7-178, PMID 20678232, PMC 2922188 (freier Volltext).
- ↑ A. M. Idris, M. S. Shahid, R. W. Briddon, A. J. Khan, J. K. Zhu, J. K. Brown: An unusual alphasatellite associated with monopartite begomoviruses attenuates symptoms and reduces betasatellite accumulation. In: J. Gen. Virol. 92. Jahrgang, Pt 3, März 2011, S. 706–717, doi:10.1099/vir.0.025288-0, PMID 21084498 (microbiologyresearch.org [PDF]).
- ↑ M. S. Nawaz-Ul-Rehman, N. Nahid, S. Mansoor, R. W. Briddon, C. M. Fauquet: Post-transcriptional gene silencing suppressor activity of two non-pathogenic alphasatellites associated with a begomovirus. In: Virology. 405. Jahrgang, Nr. 2, September 2010, S. 300–308, doi:10.1016/j.virol.2010.06.024, PMID 20598726.
- ↑ Lina Katul, Edgar Maiss and H. Josef Vetten: Sequence analysis of a faba bean necrotic yellows virus DNA component containing a putative replicase gene. In: J. Gen. Virol. 76. Jahrgang, Pt 2, 1. Februar 1995, S. 475–479, doi:10.1099/0022-1317-76-2-475, PMID 7844570 (microbiologyresearch.org [PDF]).
- ↑ Lina Katul, T. Timchenko, B. Gronenborn, H. J. Vetten: Ten distinct circular ssDNA components, four of which encode putative replication-associated proteins, are associated with the faba bean necrotic yellows virus genome. In: J. Gen. Virol. 79. Jahrgang, Pt 12, Dezember 1998, S. 3101–3109, doi:10.1099/0022-1317-79-12-3101, PMID 9880028 (microbiologyresearch.org [PDF]).
- ↑ Y. Sano, M. Wada, Y. Hashimoto, T. Matsumoto, M. Kojima: Sequences of ten circular ssDNA components associated with the milk vetch dwarf virus genome. In: J. Gen. Virol. 79. Jahrgang, Pt 12, Dezember 1998, S. 3111–3118, doi:10.1099/0022-1317-79-12-3111, PMID 9880029 (microbiologyresearch.org [PDF]).
- ↑ Rob W. Briddon, Simon E. Bull, Imran Amin, et al.: Diversity of DNA 1: a satellite-like molecule associated with monopartite begomovirus-DNA beta complexes. In: Virology. 324. Jahrgang, Nr. 2, Juli 2004, S. 462–474, doi:10.1016/j.virol.2004.03.041, PMID 15207631.
- ↑ Muhammad Shafiq Shahid, Liaqat Ali, Saiqa Andleeb: The function of the a-rich region of the alphasatellite associated with the cotton leaf curl disease in Pakistan. In: EurAsia J BioSci. 3. Jahrgang, Dezember 2009, S. 152–156 (ejobios.com (Memento des vom 8. März 2014 im Internet Archive) [abgerufen am 25. Juni 2021]). Web-Archiv vom 8. Mai 2014, doi:10.5053/ejobios.2009.3.0.19
- ↑ G. Romay, D. Chirinos, F. Geraud-Pouey, C. Desbiez: Association of an atypical alphasatellite with a bipartite New World begomovirus. In: Arch. Virol. 155. Jahrgang, Nr. 11, November 2010, S. 1843–1847, doi:10.1007/s00705-010-0760-7, PMID 20665058.
- ↑ J. Kumar, A. Kumar, J. K. Roy, R. Tuli, J. A. Khan: Identification and molecular characterization of begomovirus and associated satellite DNA molecules infecting Cyamopsis tetragonoloba. In: Virus Genes. 41. Jahrgang, Nr. 1, August 2010, S. 118–125, doi:10.1007/s11262-010-0482-7, PMID 20405195.
- ↑ I. Amin, K. Hussain, R. Akbergenov et al.: Suppressors of RNA silencing encoded by the components of the cotton leaf curl begomovirus-betasatellite complex. In: Mol. Plant Microbe Interact. 24. Jahrgang, Nr. 8, August 2011, S. 973–983, doi:10.1094/MPMI-01-11-0001, PMID 21751853.
- ↑ Chang-jun Huang, Tong Zhang, Fang-fang Li, Xin-yue Zhang, Xue-ping Zhou: Development and application of an efficient virus-induced gene silencing system in Nicotiana tabacum using geminivirus alphasatellite. In: Journal of Zhejiang University SCIENCE B. 12. Jahrgang, Nr. 2, 1. Februar 2011, S. 83–92, doi:10.1631/jzus.B1000157, PMID 21265040, PMC 3030953 (freier Volltext).
- ↑ a b Darius Kazlauskas, Arvind Varsani, Mart Krupovic: Pervasive Chimerism in the Replication-Associated Proteins of Uncultured Single-Stranded DNA Viruses, in: MDPI Viruses, Band 10, Nr. 4, Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis, 10. April 2018, 187, doi:10.3390/v10040187; siehe auch Fig. S1 in Supplement S1 (ZIP: pdf, xlsx)
- ↑ Lele Zhao, Karyna Rosario, Mya Breitbart, Siobain Duffy: Chapter Three - Eukaryotic Circular Rep-Encoding Single-Stranded DNA (CRESS DNA) Viruses: Ubiquitous Viruses With Small Genomes and a Diverse Host Range, in: Advances in Virus Research, Band 103, 2019, S. 71–133, doi:10.1016/bs.aivir.2018.10.001, Epub 5. Dezember 2018. Insbes. siehe Fig. 3
- ↑ Virus Taxonomy: 2020 Release. International Committee on Taxonomy of Viruses (ICTV), März 2021, abgerufen am 23. Mai 2021.
















