Informatics Educational Institutions & Programs
| „Idotea-Virus IWaV278“ | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||
| ||||||||||
| Taxonomische Merkmale | ||||||||||
| ||||||||||
| Wissenschaftlicher Name | ||||||||||
| „Idotea virus IWaV278“ | ||||||||||
| Kurzbezeichnung | ||||||||||
| IWaV278 | ||||||||||
| Links | ||||||||||
„Idotea-Virus IWaV278“ (Idotea wosnesenskii associated virus 278, IWaV-278) ist die vorgeschlagene Bezeichnung für eine Spezies (Art) von Viren mit einem Genom aus einer zyklischen Einzelstrang-DNA (ssDNA). Das Genom hat das für Mitglieder des Phylums Cressdnaviricota typische REP-Gen und Haarnadelstruktur (englisch stem loop), verantwortlich für den Replikationsstart.[2][3][4][5] „Idotea-Virus IWaV278“ ist vergesellschaftet (assoziiert) mit Asseln der Spezies Idotea wosnesenskii (Valvifera), die im Intertidalbereich (Litoral) der US-Pazifikküste vorkommen.[2] IWaV278 teilt Sequenzähnlichkeit und genomische Merkmale mit den Tombusviridae (ssRNA) und Circoviridae (ssDNA) und wurde der vorgeschlagenen Familie „Cruciviridae“ zugeordnet, die eine vermutete Klade chimärer Viren (genauer: DNA-RNA-Hybridviren) umfasst.
Entdeckung
Im Jahr 2017 veröffentlichten Kalia Bistolas und Kollegen eine Metagenom-Studie, in der die phylogenetischen Beziehungen zwischen neuartigen Cressdnaviricota-Genotypen (Genomen von CRESS-DNA-Viren) untersucht wurden, die mit pazifischen Asseln aus dem Intertidal assoziiert sind. Bei dem mit der Asselspezies Idotea wosnesenskii assoziierten Genotyp IWaV278 wurde dabei die bezeichnete hybride Genomzusammensetzung gefunden. Die Untersuchungen lieferten weitere Hinweise zur Klärung der Evolution einer möglichen Familie „Cruciviridae“.[2]
Genom

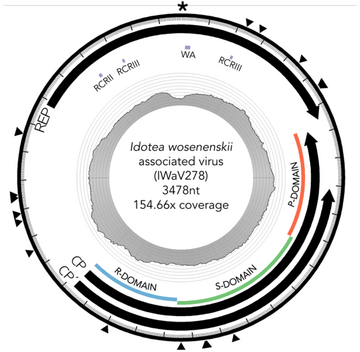
Das rechnerisch vervollständigte Genom von IWaV278 hat eine Länge von 3478 nt (Nukleotiden). Es enthält die für die virale Replikationsfähigkeit mindestens erforderlichen Offenen Leserahmen (en. open reading frames, ORFs) Rep (für das Replikations-Initiations-Protein) und Cp (auch Cap, für das Kapsidprotein).[Anm. 1] Das Kapsid-ORF von IWaV278 ist homolog zu denen, die in den (+)RNA-Viren der Familie Tombusviridae gefunden wurden. Das Rolling-Circle-Replikations-ORF Rep ist homolog zu jenen, wie sie üblicherweise in CRESS-DNA-Viren der Familie Circoviridae anzutreffen sind. Daher wird vermutet, dass IWaV278 ein neues Mitglied der vorgeschlagenen Familie „Cruciviridae“ sein könnte. Das IWaV278-Genom ist größer als bei den meisten CRESS-DNA-Viren, aber in der Größe vergleichbar mit anderen mutmaßlichen Mitgliedern der Cruciviridae/Tombusviridae-Klade (Cruciviren mit Tombusviridae-Homologie).[2][6][7][8] IWaV278 enthält eine 728 nt lange nicht-kodierende intergenische Sequenz (Zwischengensequenz oder -region, en. intergenic sequence/region, IGR) mit zwei flankierenden Ambisense-ORFs:[2]
- das Rep, homolog zum Replikase-Gen von Tadarida brasiliensis circovirus 1,[9] einem Stamm (Genotyp) der Spezies Bat associated circovirus 4 (BatACV4) der Circoviridae.
- das Cp, homolog zum „Cruciviridae“-Genotyp CRUV-15-B (AQU11701.1)[10]
Offenbar zeigt IWaV278 keine Anzeichen von Intra-Gen-Chimärismus (d. h. Anzeichen von mehrfachen Rekombinationen innerhalb von Rep) oder partiellem Rep-Gen-Austausch – im Gegensatz zu mehreren anderen zuvor bekannt gewordenen Genomen von Cruciviren, die auch Homologien zu anderen CRESS-DNA-Viren (neben Circoviridae auch Nanoviridae oder Geminiviridae) zeigen.[6][2]
Phylogenie
Kalia Bistolas et al. ermittelten 2017 die Phylogenie von IWaV278. Diese phylogenetische Analyse zeigt, dass das Rep von IWaV278 mit einer monophyletischen Klade nicht-chimären Reps homolog zu Circovirus verbunden ist, was möglicherweise eine singuläre (stammesgeschichtlich einmalige) Übernahme dieses Gens/ORFs durch einen Vorfahren der CRESS-DNA-Viren (Cressdnaviricota) anzeigt.[2]

(A) Rep (Replikations-Initiation) und (B) Cp (Kapsid).[Anm. 2][2]
Der untere Teil (B) zeigt die Verwandtschaft des Cp-Gens/ORFs. Die IWaV278 bzgl. des Cp am nächsten stehenden Vertreter sind drei Sequenzen, die bereits für die „Cruciviridae“ vorgeschlagen wurden, darunter „Boiling Springs Lake RNA-DNA Hybrid Virus“ (BSL-RDHV); aus dem Rahmen fällt jedoch „Circoviridae 14 LDMD-2013“ Weiter entfernt finden sich in diesem Baum andere Sequenzen chimärer Viren (chimeric viruses), teilweise für die „Cruciviridae“ vorgeschlagen. „RDHV-like virus SF1“ mit einer Genomlänge von 3.040 nt ist zusammen mit SF2 und SF3 nahe verwandt mit dem Stamm Marine RNA virus JP-B[11][12] der Spezies Jericarnavirus B (Gattung Locarnavirus in der Familie Marnaviridae).
Der obere Teil (A) zeigt die Verwandtschaft des Rep-Gens/ORFs. Am nächsten zu IWaV278 bzgl. des Rep steht die nicht klassifizierten Spezies „Lake Sarah-associated circular virus-42“,[13] danach die Spezies „Circoviridae 14 LDMD-2013“,[14] ein vorgeschlagenes Mitglied der Circoviridae. Offenbar gruppiert sich IWaV278 in die von Simon Roux und Kollegen 2013 identifizierte Klade der RDHVs (bzw. „Cruciviridae“) mit Circoviridae-ähnlichem Rep.[8]
Biogeographie
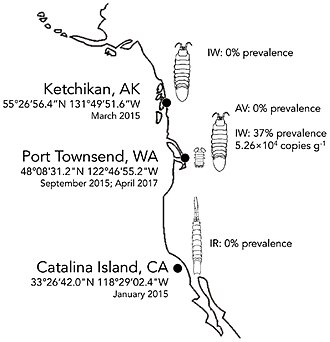
IWaV278 wurde in Idotea wosnesenskii (IW), aber weder in den verwandten Asseln Idotea resecata (IR), noch in den sympatrischen Asseln Gnorimosphaeroma oregonensis (GO) oder im primärem Nahrungssubstrat (dem Seetang Nereocystis sp.) nachgewiesen.[2]
Gegenstand der 2017 veröffentlichten Untersuchung von Kalia Bistolas und Kollegen waren die Asselarten Idotea wosnesenskii, I. resecata und Gnorimosphaeroma oregonensis. Obwohl I. wosnesenskii ein sehr weites Verbreitungsgebiet entlang der nordamerikanischen Pazifikküste hat (von Alaska über Kanada bis Zentralkalifornien, USA), wurde IWaV278 nur an einem einzigen Standort (Port Townsend, WA, USA), im Jahr 2015 gefunden. IWaV278 fehlte insbesondere an einem nördlicheren Standort (Ketchikan, AK, USA). Dies deutet darauf hin, dass die Ausbreitung dieses Genotyps geografisch durch andere Faktoren als die Verbreitung von I. wosnesenskii eingeschränkt ist. Diese Genomsequenzen waren assoziiert ausschließlich mit der Asselspezies I. wosnesenskii. Sie waren häufiger bei männlichen Exemplaren zu finden, und meist in den Strukturen des Exoskeletts. Sequenzanalysen von 18S-rRNA identifizierten zwei Protisten der Alveolata, die mit IWaV278-positivem Gewebe assoziiert waren. Vermutlich sind daher diese Epibionten von I. wosnesenskii die Wirte von IWaV278 (nicht die Asseln selbst).[2] Da bisher nur Informationen aus der Metagenomik vorliegen, können noch keine sicheren Aussagen über die Wirte von IWaV278 getroffen werden, ähnlich wie bei anderen Cruciviren.
Anmerkungen
- ↑ Es wird hier einer allgemeinen Praxis gefolgt, die Abkürzungen für Gene bzw. ORFs kursiv zu schreiben, dagegen die Abkürzungen für die kodierten Proteine in Normalschrift.
- ↑ Details zu den beiden angegebenen Kladogrammen:
(A) Rep-Phylogenie:- NCBI Human stool-associated circular virus NG13 (no rank, ADD62475.1) — ICTV: Species Human associated circovirus 1 — Gattung Circovirus — Außengruppe 1
- NCBI: Silurus glanis circovirus (no rank, AEW70742.1) — ICTV: Species European catfish circovirus — Gattung Circovirus
- NCBI: Odonata-associated circular virus-20 (species, AJD07502.1, unclassified, circular ssDNA)
- NCBI: Circoviridae 14 LDMD-2013 (species, YP_009109670.1, environmental samples) — Familie Circoviridae
- NCBI: Idotea virus IWaV278 (species, MG023125.1) — Familie „Cruciviridae“
- NCBI: Lake Sarah-associated circular virus-42 (species, YP_009237578.1, unclassified —NCBI: Isolate LSaCV-42-LSCO-2013, circular DNA)
- NCBI: Chimpanzee stool avian-like circovirus Chimp17 (species, ADD62465.1) — ICTV: Species Chimpanzee associated circovirus 1 — Gattung Circovirus
- NCBI: Canary circovirus (species, NP_573442.1) — ICTV: Species Canary circovirus — Gattung Circovirus
- NCBI: Tick circovirus (no rank, ARO77516.1) — ICTV: Species Tick associated circovirus 2 — Gattung Circovirus
- NCBI: Beak and feather disease virus (species, ACU57088.1, AGC55152.1, AGC55154.1, AKO63030.1, AKO63024.1, AKO63008.1, AFM55150.1, AKO63028.1) — ICTV: Species Beak and feather disease virus — Gattung Circovirus
- NCBI: Duck circovirus (species, AFJ74615.1) sowie NCBI: Muscovy duck circovirus (no rank, ACT79249.1, ADX06802.1) — ICTV: Species Duck circovirus — Gattung Circovirus
- NCBI: Rhinolophus ferrumequinum circovirus 1 (no rank, AFK85001.1) — ICTV: Species Bat associated circovirus 3 — Gattung Circovirus
- NCBI: Tadarida brasiliensis circovirus 1 (no rank, YP_009170674.1) — ICTV: Species Bat associated circovirus 4 — Gattung Circovirus
- NCBI: Porcine circovirus 3 (species, APC65715.1, ARD71104.1) — ICTV: Species Porcine circovirus 3 - Gattung Circovirus
- NCBI: Bat associated cyclovirus 6 (species, AIF76249.1) — ICTV: Species Bat associated cyclovirus 6 — Gattung Cyclovirus — Außengruppe 2
- Cruciviridae sp. NCBI: AQU11706.1 (Isolat CRUV-16-B) — nach Quaiser et al. (2016) „Crucivirus-16-B“ - Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11708.1 (Isolat CRUV-17-B) — nach Quaiser et al. (2016) „Crucivirus-17-B“ - Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11716.1 (Isolat CRUV-19-B) — nach Quaiser et al. (2016) „Crucivirus-19-B“ - Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11721.1 (Isolat CRUV-21-B) — nach Quaiser et al. (2016) „Crucivirus-21-B“ - Familie „Cruciviridae“
- NCBI: Hubei narna-like virus 10 (species, unclassified RNA viruses ShiM-2016) — Phylum Lenarviricota? — Realm Riboviria
- NCBI: Changjiang narna-like virus 2 (species, unclassified RNA viruses ShiM-2016) — Phylum Lenarviricota? — Realm Riboviria
- NCBI: YP_009142777.1 verweist auf Chimeric virus 14 (species, CHIV14) — CHIV4 ist offenbar ein Verschreiber, vgl. Krupovic et al. (2015)
- Cruciviridae sp. NCBI: AQU11701.1 (Isolat CRUV-15-B) — nach Quaiser et al. (2016) „Crucivirus-15-B“ — Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11727.1 (Isolat CRUV-24-F) — nach Quaiser et al. (2016) „Crucivirus-24-F“ — Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11725.1 (Isolat CRUV-23-F) — nach Quaiser et al. (2016) „Crucivirus-23-F“ — Familie „Cruciviridae“
- NCBI: RDHV-like virus SF1 (species, AKM12421.1) — San Francisco wastewater, „RDHV-like“ heißt vielleicht „Cruciviridae“, vermutlich aber einfach nur Cressdnaviricota
- uncultured chimeric virus NCBI: AOV86298.1 — Isolat Wastewater_Circular_Virus_FL44 Tallahassee
- uncultured chimeric virus NCBI: AOV86314.1 — Isolat Wastewater_Circular_Virus_FL52 Tallahassee
- Cruciviridae sp. NCBI: AQU11772.1 (Isolat CRUV-40-B) — nach Quaiser et al. (2016) „Crucivirus-40-B“ — Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11775.1 (Isolat CRUV-41-F) — nach Quaiser et al. (2016) „Crucivirus-41-F“ — Familie „Cruciviridae“
- NCBI: Idotea virus IWaV278 (species, MG023125.1) - Familie „Cruciviridae“
- NCBI: Circoviridae 2 LDMD-2013 (species, YP_009109631.1, environmental samples) — Familie Circoviridae
- NCBI: Boiling Springs Lake RNA-DNA hybrid virus (species, YP_009094499.1, environmental samples, unclassified virus) — „Boiling Springs Lake RNA-DNA Hybrid Virus“ (BSL-RDHV) ist aber erstes der BSL-RDHV-like viruses, d. h. Typus seiner Klade in dieser Gruppe, und diese wurde vorgeschlagen als Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11796.1 (Isolat CRUV-49-B) — nach Quaiser et al. (2016) „Crucivirus-49-F“ — Familie „Cruciviridae“
- Cruciviridae sp. NCBI: AQU11800.1 (Isolat CRUV-50-F) — nach Quaiser et al. (2016) „Crucivirus-50-F“ — Familie „Cruciviridae“
- NCBI: Tomato yellow margin leaf curl virus (species, YP_006456.1) — ICTV: Spezies Tomato yellow margin leaf curl virus — Gattung Begomovirus — Familie Geminiviridae — Phylum Cressdnaviricota — Außengruppe
Literatur
- Achim Quaiser, Mart Krupovic, Alexis Dufresne, André-Jean Francez, Simon Roux: Diversity and comparative genomics of chimeric viruses in Sphagnum-dominated peatlands, in: Virus Evolution, Band 2, Nr. 2, Juli/Oktober 2016, vew025, doi:10.1093/ve/vew025.
F=Fenn: Nieder- oder Flachmoor (en. Fen),
B=(Hoch-)Moor (en. Bog) - Mart Krupovic, Ning Zhi, Jungang Li, Gangqing Hu, Eugene V. Koonin, Susan Wong, Sofiya Shevchenko, Keji Zhao, Neal S. Young: Multiple Layers of Chimerism in a Single-Stranded DNA Virus Discovered by Deep Sequencing, in: Genome Biol. Evol. (GBE) Band 7, Nr. 4, 16. Februar 2015, S. 993–1001, doi:10.1093/gbe/evv03 — CHIV14
Einzelnachweise
- ↑ a b ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ↑ a b c d e f g h i j k l Kalia S. I. Bistolas, Ryan M. Besemer, Lars G. Rudstam, Ian Hewson: Distribution and Inferred Evolutionary Characteristics of a Chimeric ssDNA Virus Associated with Intertidal Marine Isopods, in: MDPI Viruses Band 9, Nr 12, 361; Special Issue Viral Recombination: Ecology, Evolution and Pathogenesis; Dezember 2017, Epub 26. November 2017; doi:10.3390/v9120361, PMID 29186875, PMC 5744136 (freier Volltext).
- ↑ NCBI: Idotea virus IWaV278 (species)
- ↑ UniProt: Idotea virus IWaV278 (SPECIES)
- ↑ Pfam: Proteome: Idotea virus IWaV278 (NCBI tax. ID 2058759), Memento im Webarchiv vom 10. Juni 2021
- ↑ a b Mart Krupovic, Ning Zhi, Jungang Li, Gangqing Hu, Eugene V. Koonin, Susan Wong, Sofiya Shevchenko, Keji Zhao, Neal S. Young: Multiple Layers of Chimerism in a Single-Stranded DNA Virus Discovered by Deep Sequencing, in: Genome Biol. Evol. (GBE) Band 7, Nr. 4, 16. Februar 2015, S. 993–1001, doi:10.1093/gbe/evv03
- ↑ Achim Quaiser, Mart Krupovic, Alexis Dufresne, André-Jean Francez, Simon Roux: Diversity and comparative genomics of chimeric viruses in Sphagnum-dominated peatlands, in: Virus Evolution, Band 2, Nr. 2, Juli/Oktober 2016, vew025, doi:10.1093/ve/vew025
- ↑ a b Simon Roux, François Enault, Gisèle Bronner, Daniel Vaulot, Patrick Forterre, Mart Krupovic: Chimeric viruses blur the borders between the major groups of eukaryotic single-stranded DNA viruses, in: Nat Commun 4:2700, 6. November 2013, doi:10.1038/ncomms3700. Siehe insbesondere Supplement 2 (xls).
- ↑ NCBI: Tadarida brasiliensis circovirus 1 (no rank)
- ↑ NCBI: capsid protein [Cruciviridae sp.] GenBank: AQU11701.1
- ↑ Alexander L. Greninger, Joseph L. DeRisi: Draft Genome Sequences of Marine RNA Viruses SF-1, SF-2, and SF-3 Recovered from San Francisco Wastewater, in: Genome Announc. 3(3), Mai-Juni 2015, Epub 18. Juni 2015, e00653-15 doi:10.1128/genomeA.00653-15, PMC 4472900 (freier Volltext), PMID 26089423
- ↑ NCBI: Marine RNA virus JP-B (no rank)
- ↑ NCBI: Lake Sarah-associated circular virus-42 (species)
- ↑ NCBI: Circoviridae 14 LDMD-2013 (species), siehe auch NCBI: LDMD-2013 (list)
















